Hasta mediados del siglo 20 no se sospechaba que el ácido disoxirribonucleico, ADN, fuera la molécula capaz de asegurar la transmisión de los caracteres hereditarios de célula a célula, generación tras generación. Su limitada variedad química no permitía suponer que poseyera la versatilidad y ductilidad necesarias para almacenar la información genética de los seres vivos.
En 1869 un biólogo suizo Johann Friedrich Miesscher, utilizo primero alcohol caliente y luego una pepsina enzimatica, que separa la membrana celular y el citoplasma de la célula, el científico quería aislar el núcleo celular, concretamente en los núcleos de las células del pus obtenidas de los vendajes quirúrgicos desechados y en la esperma del salmón, sometió a este material a una fuerza centrifuga para aislar a los núcleos del resto y luego sometió solo a los núcleos a un análisis químico.
De esta manera Miescher identifico a un nuevo grupo de substancias celulares a las que denomino nucleínas, observo la presencia de fósforo, luego Richard Altmann las identifico como ácidos y les dio el nombre de ácidos nucleicos.
Robert Feulgen, en 1914, describió un método para revelar por tinción el ADN, basado en el colorante fucsina. Se encontró, utilizando este método, la presencia de ADN en el núcleo de todas las células eucariotas, específicamente en los cromosomas.
Durante los años 20, el bioquímico P.A. Levene analizo los componentes del ADN, los ácidos nucleicos y encontró que contenía cuatro bases nitrogenadas: citosina y timina (pirimidinas), adenina y guanina (purinas); el azúcar desoxirribosa; y un grupo fosfato. También demostró que se encontraban unidas en el orden fosfato-azúcar-base, formando lo que denomino un nucleótido. Levene también sugirió que los nucleótidos se encontraban unidos por los fosfatos formando el ADN. Sin embargo, Levene pensó que se trataban de cadenas cortas y que las bases se repetían en un orden determinado.
En el año 1928 Frederick Griffith investigando una enfermedad infecciosa mortal, la neumonía, estudió las diferencias entre una cepa de la bacteria Streptococcus peumoniae que producía la enfermedad y otra que no la causaba. La cepa que causaba la enfermedad estaba rodeada de una cápsula (también se la conoce como cepa S, del ingles smooth, o sea lisa, que es el aspecto de la colonia en las placas de Petri). La otra cepa (la R, de rugosa, que es el aspecto de la colonia en la placa de Petri) no tiene cápsula y no causa neumonía.
Griffith inyectó las diferentes cepas de la bacteria en ratones. La cepa S mataba a los ratones mientras que la cepa R no lo hacía. Luego comprobó que la cepa S, muerta por calentamiento, no causaba neumonía cuando se la inyectaba. Sin embargo cuando combinaba la cepa S muerta por calentamiento, con la cepa R viva, es decir con componentes individuales que no mata a los ratones e inyectaba la mezcla a los ratones, los ratones contraían la neumonía y morían.
Las bacterias que se aislaban de los ratones muertos poseían cápsula y, cuando se las inyectaba, mataban otros ratones. Frederick Griffith fue capaz de inducir la transformación de una cepa no patogénica Streptococcus pneumoniae en patogénica. Griffith postuló la existencia de un factor de transformación como responsable de este fenómeno.
1 . 1. 3 El descubrimiento del codigo genetico
Cuando James Watson, Francis Crick, Maurice Wilkins y Rosalind Franklin descubrieron la estructura del ADN, se comenzó a estudiar en profundidad el proceso de traducción en las proteínas. En 1955, Severo Ochoa y Marianne Grunberg-Manago aislaron la enzima polinucleótido fosforilasa, capaz de sintetizar ARNm sin necesidad de modelo a partir de cualquier tipo de nucleótidos que hubiera en el medio. Así, a partir de un medio en el cual tan sólo hubiera UDP (urdín difosfato) se sintetizaba un ARNm en el cual únicamente se repetía el ácido urídico, el denominado poli-U (....UUUUU....). George Gamow postuló que un código de codones de tres bases debía ser el empleado por las células para codificar la secuencia aminoacídica, ya que tres es el número entero mínimo que con cuatro bases nitrogenadas distintas permiten más de 20 combinaciones (64 para ser exactos).
Los codones constan de tres nucleótidos fue demostrado por primera vez en el experimento de Crick, Brenner y colaboradores. Marshall Nirenberg y Heinrich J. Matthaei en 1961 en los Institutos Nacionales de Salud descubrieron la primera correspondencia codón-aminoácido. Empleando un sistema libre de células, tradujeron una secuencia ARN de poli-uracilo (UUU...) y descubrieron que el polipéptido que habían sintetizado sólo contenía fenilalanina. De esto se deduce que el codón UUU específica el aminoácido fenilalanina. Continuando con el trabajo anterior, Nirenberg y Philip Leder fueron capaces de determinar la traducción de 54 codones, utilizando diversas combinaciones de ARNm, pasadas a través de un filtro que contiene ribosomas. Los ARNt se unían a tripletes específicos.
Posteriormente, Har Gobind Khorana completó el código, y poco después, Robert W. Holley determinó la estructura del ARN de transferencia, la molécula adaptadora que facilita la traducción. Este trabajo se basó en estudios anteriores de Severo Ochoa, quien recibió el premio Nobel en 1959 por su trabajo en la enzimología de la síntesis de ARN. En 1968, Khorana, Holley y Nirenberg recibieron el Premio Nobel en Fisiología o Medicina por su trabajo.
1 . 1. 4 El modelo de oparen
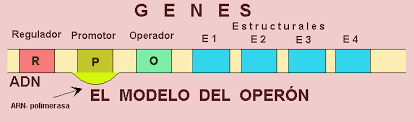
Un Operón se define como una unidad genética funcional formada por un grupo o complejo de genes capaces de ejercer una regulación de su propia expresión por medio de los sustratos con los que interaccionan las proteínas codificadas por sus genes. Este complejo está formado por genes estructurales que codifican para la síntesis de proteínas (generalmente enzimas), que participan en vías metabólicas cuya expresión generalmente está regulada por otros 3 factores de control, llamados:
- Factor promotor: zona que controla el inicio de la transcripción del operón, ya que la ARN Polimerasa tiene afinidad por ella. Realmente, como un gen es cada unidad de transcripción independiente, y puesto que el operón tiene un único promotor que controla toda su expresión, no hay elementos para decir que se trate de "varios genes" de expresión coordinada; más correcto sería decir que el operón es un único gen que codifica un ARNm policistrónico (es decir, con muchos codones de inicio y paro, con lo que a la hora de traducirse dará lugar a varias proteínas independientes). Sin embargo, es común referirse a los "genes" del operón para hacer referencia a las regiones que, una vez transcritas, codificarán proteínas independientes.
- Operador: zona de control que permite la activación/desactivación del promotor a modo de "interruptor génico" por medio de su interacción con un compuesto inductor. Esto lo logra porque tiene secuencias reconocibles por proteínas reguladoras. Tras su unión, por plegamientos tridimensionales interacciona con la zona del promotor, donde las proteínas reguladoras que se han unido contactan con la ARN Polimerasa, aumentando o disminuyendo su afinidad por el promotor, y con ello dando lugar a la expresión/represión del resto de los genes estructurales.
- Gen regulador: alguno de los genes del operón pueden codificar factores de transcripción que se unan al promotor, regulando así la propia expresión del operón. A toda regulación de la expresión realizada desde dentro del gen u operón se le llama "regulación en cis", pero puede haber también genes muy alejados del operón que codifiquen factores de transcripción para uno o varios otros genes u operones, y en este caso se hablaría de "regulación en trans".
1. 2 La biologia molecular en mexico
La biología molecular nace, asimismo, de la bioquímica. La bioquímica en sí, se gestó dentro del pensamiento cuantitativo, particularmente con la visión de que la vida se podía explicar a través de una serie de reacciones químicas, catalizadas por enzimas. Así se construyeron los grandes esquemas de las vías metabólicas que incluyen, entre otros muchos, el ciclo de Krebs, el ciclo de la urea, la cadena respiratoria, la biosíntesis de ácidos grasos, de las hormonas, y de las vitaminas y la fotosíntesis. Como en todo el mundo, la biología molecular en México nació de la bioquímica. Hasta principios de la década de 1970.
FORTALEZAS
En el periodo entre 1996 y 2006 mexico produjo el 0 . 554 de la ciencia mundial , considerada como articulos publicos en revistas indexadas.
Mexico cuenta con sociedad mexicana de la bioquimica (SMB) fundada en 1957.
Cuenta con 538 socios correspondientes a un numero subestimado de investigadores en el area.
Las investigaciones en mexico se llevan a cabo principalmente en la (UNAM) con un 50% de investigacion cientifica en el pais.
En (CONACYT) constituye la fuente principal para la investigacion.
Mexico ocupoa el tercer lugar en la region latinoamericana despues de brazil y argentina.
DEBILIDADES
México se encuentra muy por debajo de la media en cuanto a la elaboración de artículos científicos.
La eliminacion de apoyo para infraestructuras, que permiten renovar latentamente los equipos viejos o obsoletos.
México se encuentra muy por debajo de la media en cuanto a la elaboración de artículos científicos.
Las inconsistencias en el programa de repartición de científicos Mexicanos que se encuentran en el extranjero.













